Proteine sind derzeit selbst vielen Laien ein Begriff: So ist der Stachel des Coronavirus auch als "Spike-Protein" bekannt, und der Protein-Impfstoff von Novavax wurde kürzlich in Europa zugelassen. Doch auch sonst gehören Proteine zum Alltag: in der Nahrung oder in Form von Enzymen, die den Stoffwechsel steuern.
"Proteine, Proteine überall" überschreibt auch Holden Thorp einen Leitartikel des Wissenschaftsmagazins Science. Als Chefredakteur des Journals kürt Thorp einen Algorithmus zum "Durchbruch des Jahres 2021", der bald eine riesige Wissenslücke in der Erforschung von Proteinen schließen könnte: Die dreidimensionale Form der meisten Proteine im Tier- und Pflanzenreich ist noch unbekannt, da es lange dauert, sie experimentell zu bestimmen.
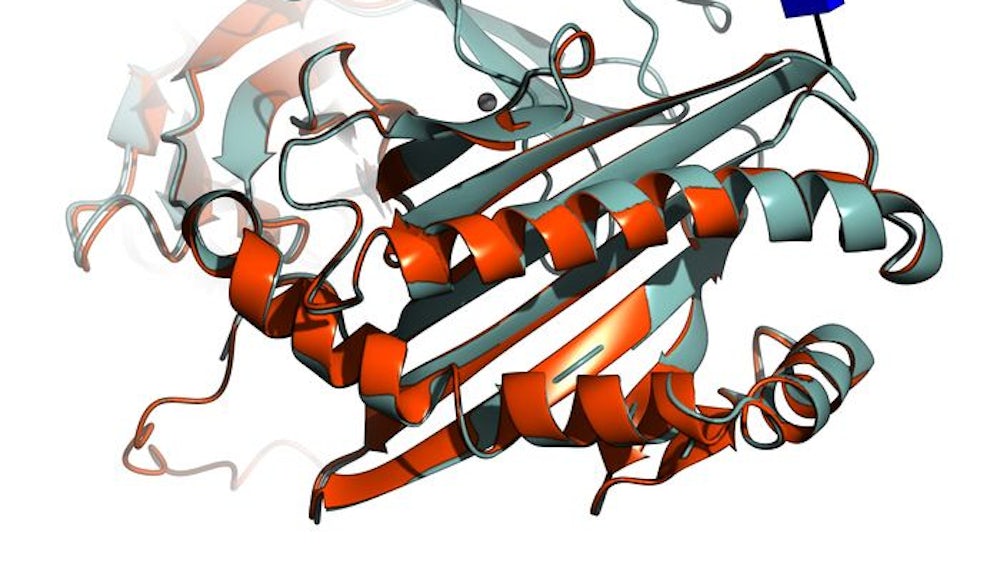
Eine künstliche Intelligenz namens Alphafold hingegen berechnet die 3-D-Strukturen rein virtuell. Das hat die Software der britischen Google-Schwester Deepmind inzwischen mit allen Proteinen des menschlichen Körpers getan. 2022 soll die KI die 3-D-Formen sämtlicher rund 100 Millionen der Biologie bekannten Proteine fertigstellen.
"Alphafold erleichtert uns das Leben ungemein", sagt Christian Löw vom Europäischen Laboratorium für Molekularbiologie in Hamburg. Die KI wirkt wie ein Turbo für die Biowissenschaften, die nun Proteine schneller erforschen und in der Folge schneller neue Wirkstoffe finden können.
Warum sind Proteine so formenreich? Proteine sind Ketten aus Hunderten Einzelbestandteilen, sogenannten Aminosäuren. Der menschliche Körper nutzt 21 verschiedene Aminosäuren. So ergibt sich eine Unzahl von Kombinationsmöglichkeiten, genannt "Sequenzen". Da verschiedene Aminosäuren sich anziehen oder abstoßen, faltet sich die Kette zu einer komplexen Figur. Diese dreidimensionale Form eines Proteins bestimmt seine Funktion. Es bildet zum Beispiel Bindetaschen aus, in die nur ganz bestimmte Wirkstoffe passen, wie ein Schlüssel ins Schloss. Passt der Schlüssel, dann führt das Protein seine Funktion aus, transportiert etwa ein angedocktes Nährstoffmolekül in das Innere einer Zelle.
Das Wechselspiel innerhalb von Proteinen ist derart komplex, dass selbst Supercomputer kapitulieren
Um die Funktion zu verstehen, müssen Forscher daher die Gestalt der molekularen Tausendsassas kennen. Doch Proteine sind submikroskopisch klein. Ihre Form lässt sich nur mit aufwendigen Verfahren wie der Elektronenmikroskopie bestimmen - und das nicht immer erfolgreich. Die Sequenz hingegen lässt sich leicht analysieren. Also liegt der Versuch nahe, direkt aus der Sequenz per Computer die Form zu berechnen. Doch das physikalische und chemische Wechselspiel zwischen den einzelnen Aminosäuren ist derart komplex, dass selbst Supercomputer kapitulieren.
Alphafold geht die Sache anders an. Die Software setzt auf Mustererkennung. Dazu nutzt sie neuronale Netze. Diese Art von KI lernt durch Training mit Bildern bekannten Inhalts, auf neuen Bildern ähnliche Bildinhalte zu erkennen, etwa Gesichter oder Autos.
Auch Proteinsequenzen zeigen Muster, die Alphafold ausnutzt. In verschiedenen Spezies bilden ähnliche Sequenzen ähnliche 3-D-Strukturelemente aus. Solche und andere Muster hat die KI durch das Training an bekannten Proteinstrukturen erlernt, die in einer zentralen Datenbank offen verfügbar sind. Mit großem Erfolg: Alphafold sagt unbekannte Formen teilweise mit atomgenauer Präzision voraus.
"Alphafold hat unsere Arbeitsweise revolutioniert", sagt der Biochemiker Christian Löw. Sein Team untersucht ein Protein namens PepT1, das in der Zellmembran von Darmzellen sitzt und Nährstoffe sowie Medikamente, etwa Blutdrucksenker, in die Zellen transportiert. Es wirkt wie eine Schleuse, die sich zunächst nach außen öffnet, das Transportgut aufnimmt und es dann ins Zellinnere entlässt. "Den nach innen geöffneten Zustand kannten wir noch nicht", sagt Löw. Alphafold hat ihn vorhergesagt. Der Vorteil: "Wenn man PepT1 versteht, kann man Medikamente so designen, dass sie effizienter in die Blutbahn aufgenommen werden." Eine kleinere Dosis könnte dann den gleichen Effekt haben.
"Früher dauerte es eine Doktorarbeit lang, um die 3-D-Struktur eines Proteins herauszufinden", erzählt Löw. Alphafold habe nur wenige Stunden gebraucht. "Jetzt können wir mit einem Strukturmodell beginnen und dadurch früh Hinweise auf die Funktionsweise eines Proteins erhalten."
Die Dynamik von Proteinen kann nicht einmal Alphafold abbilden
Löws Kollege Jan Kosinski gibt ihm recht. Der Forscher untersucht einen Komplex aus Hunderten Proteinen, der eine Pore in der Wand von Zellkernen bildet. Bis vor Kurzem kannten die Forscher nur einen kleinen Teil der Proteine, dank Alphafold sind nun die Formen eines Großteils bekannt. "Das hätte normalerweise bis zu zehn Jahre gedauert und viele Forschende beschäftigt", sagt Kosinski. Alphafold verwandele das ganze Forschungsfeld. "Es dominiert die Diskussionen auf Konferenzen", sagt Kosinski.
Alphafold werde die experimentellen Methoden nicht ersetzen, betont Löw. Denn die Software hat ihre Grenzen. Einen wichtigen biologischen Faktor ignoriert die Software ganz: die Zeit. "Proteine sind aber sehr dynamisch", erklärt Löw. Sie verformen sich, wenn sie sich mit anderen Proteinen zu einem größeren Komplex verbinden oder wenn sich ein Wirkstoff anlagert. Manche Proteine hätten "Schwänze", die flexibel seien "wie Spaghetti", ergänzt Kosinski. Daher bleibe Alphafolds Modell der Kernporen unvollständig, räumt der Forscher ein.
Ein Manko ist auch die schwankende Präzision, mit der Alphafold die Position einzelner Atome innerhalb eines Proteins angibt. Die sei ausgerechnet an den Bindetaschen oft gering, sagt Christofer Tautermann, Leiter der Computerchemie beim Pharmaunternehmen Boehringer Ingelheim. Tautermann lotet aus, ob Alphafold die aufwendige Suche nach neuen Wirkstoffen erleichtern kann. "Der Kandidat muss perfekt in die Tasche passen", erläutert der Chemiker und Mathematiker. Er sei begeistert von Alphafold, sagt Tautermann, aber die Software sei kein Wundermittel. Doch er ist zuversichtlich: "Mit mehr Trainingsdaten von experimentell untersuchten Bindetaschen könnte eine KI diese mit zuverlässiger Präzision vorhersagen."